
|
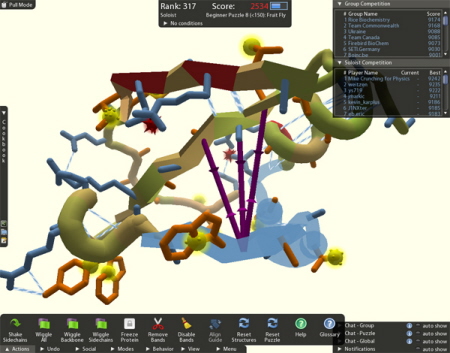
« Science Komputerowi gracze rozwiązują trudne problemy w badaniach nad AIDS Translation: Justyna Trawińska Podczas gdy naukowcy borykają się z problemem od ponad dziesięciu lat, kilku z nich pomyślało: „Wiemy co trzeba zrobić! Poprosimy o pomoc graczy komputerowych." To właśnie zrobił Firas Khatib z uniwersytetu waszyngtońskiego. Wynik: on i jego legion grających współautorów przełamali długotrwały problem w badaniach na temat AIDS, nad którym naukowcy zastanawiali się od lat. Im zajęło to trzy tygodnie. Ochotnicy Khatib'a grali w Foldit — program, który stawiał szatańskie wyzwania naukowe tak jak konkurencyjne wieloosobowe gry komputerowe. Sięga on do umiejętności zbiorowego rozwiązywania problemów dziesiątków tysięcy ludzi, z których większość ma niewielkie lub żadne zaplecze naukowe. Oto, co napisałem o Foldit w ubiegłym roku:
W ubiegłym roku Cooper udowodnił, że gracze Foldit byli lepsi w rozwiązywaniu wielu struktur białkowych od programu Rosetta. Używali szerokiego zakresu strategii, potrafili wybrać najlepsze miejsce by zacząć próby i byli lepsi w długoterminowym planowaniu. Ludzka intuicja dysponuje umiejętnością mechanicznego rozwiązywania zadań matematycznych. W tym roku Khatib chciał zobaczyć, czy społeczność Foldit, mogłaby rozwiązać nowsze problemy. Zapisał graczy na odbywający się dwa razy do roku konkurs nazwany CASP (Critical Assessment of Techniques for Protein Structure Prediction, w wolnym tłumaczeniu: Krytyczne Oceny Technik Przewidywania Struktur Białek), gdzie biolodzy strukturalni z całego świata konkurowali w przewidywaniu struktur białkowych, które zostały prawie rozwiązane. Na początek dostawali najlepsze prognozy z programu Rosetta. Potem byli zdani sami na siebie. Gracze Khatib'a, mający nazwy takie jak Foldit Contenders Group oraz Foldit Void Crushers Group, mieli różnego rodzaju sukcesy w konkursie. W wielu kategoriach poszło im w miarę dobrze, lecz nie mogli prześcignąć najlepszych grup. Nie byli tak dobrzy w używaniu struktur podobnych białek by podrasować je do tych, nad którymi pracowali. Czasami brnęli w ślepy zaułek, jeśli zaczęli w złym miejscu. Pod jednym względem jednak ich strategia, usprawniania struktur wyjściowych do najdoskonalszych jak tylko to możliwe, doprowadziła do jednego z „najbardziej spektakularnych sukcesów" tego konkursu. W większości jednak za bardzo skupiali się na podrasowywaniu niedoskonałych jeszcze rozwiązań, podczas gdy inne drużyny osiągnęły lepsze rezultaty poprzez wprowadzanie zmian na większą skalę. Wyciągając wnioski z tej lekcji Khatib zrobił eksperyment. Poruszył on losowo wyjściowe struktury białek, by stworzyć szeroką gamę złych odpowiedzi, które mogły z kolei być udoskonalane przez graczy. Podczas swoich zmagań zaproponowali najlepsze możliwe odpowiedzi dla większości najtrudniejszych wyzwań w konkursie. To był sukces, a więcej sukcesów rysuje się na horyzoncie. Po zawodach gracze rozwiązali jeszcze ważniejszy problem. Odkryli strukturę białka należącego do małpiego wirusa Mason-Pfizer (M-PMV), blisko spokrewnioną z wirusem HIV, który powoduje u małp AIDS. Wirusy te tworzą wiele swoich białek w jednym bloku. Muszą być pocięte na fragmenty, a używają do tego enzymatycznych nożyczek — proteaz. Wielu naukowców próbuje znaleźć leki, które wyłączą proteazy. Jeśli one nie działają, to wirus jest ujarzmiony — jest jak mechanik, który nie może wyjąć żadnego ze swoich narzędzi ze skrzynki. By wyłączyć proteazę wirusa M-PMV, musimy wiedzieć jak dokładnie wygląda. Tak jak prawdziwe nożyczki, proteazy składają się z dwóch połówek, które muszą zamykać się by wykonały swoją pracę. Gdybyśmy wiedzieli gdzie połówki łączą się ze sobą, moglibyśmy stworzyć leki przeciwdziałające ich łączeniu się. Do tej chwili, naukowcy byli tylko w stanie rozpoznać dwie połówki razem. Spędzili bezowocnie ponad dziesięć lat próbując odkryć strukturę pojedynczo wyizolowanej połówki. Gracze Foldit nie mieli takich problemów. Zaproponowali wiele odpowiedzi, każda z nich była bliska perfekcji. W kilka dni Khatib udoskonalił ich rozwiązania, by ustalić ostateczną strukturę białka, i już zauważył cechy, które mogą być przydatne dla nowych leków.
Uzupełnienie: Stephen Curry, który pracuje nad strukturami białek, w recenzji tej publikacji napisał: „Zasługą, którą należy podkreślić, jest z pewnością innowacyjne podejście do problemu określenia krystalicznych struktur białek. Poza tym podoba mi się idea 'obywatela nauki'. Pytanie w jakim stopniu gracze rozumieją naukę, ale zaangażowanie w tego rodzaju badaniach, nawet jeśli jest tylko na poziomie komputerowej gry, jest niewątpliwie dobrą rzeczą." Curry podkreśla, że struktura dla tego białka, ustalona przy użyciu innej metody zwanej magnetycznym rezonansem jądrowym, była już opublikowana w 2003, . Khatib odpowiada, że nie jest to „dokładnie tak", i że gracze usiłowali wykorzystać wcześniejsze rezultaty dla dalszego postępu. Curry utrzymuje, iż nie ma tu szczegółowego opisu różnic między wcześniejszymi strukturami i tymi nowymi. Ponadto, Khatib nie precyzuje jak blisko spokrewniona jest proteaza M-PMV z proteazą HIV. " Ta informacja ma zasadnicze znaczenie dla podjęcia decyzji, czy struktura proteazy M-PMV będzie w jakikolwiek sposób wykorzystana, jako szablon w projektowaniu nowej klasy leków skierowanych przeciwko proteazie HIV. Jeśli artykuł ten będzie recenzowany, proszę o uwzględnienie tych informacji, ponieważ są niezbędne do tego, by obserwowane różnice w strukturze miały sens, " pisze Khatib. Not Exactly Rocket Science/Discover, 18 września 2011r.
« Science (Published: 21-11-2011 )
page 7552 |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| [ Cooperation ] [ Advertise ] [ Map of the site ] [ F.A.Q. ] [ Store ] [ Sign up ] [ Contact ] The Rationalist © Copyright 2000-2018 (English section of Polish Racjonalista.pl) | ||



 Mieszka w Londynie i pracuje w Cancer Research UK. Jego blog „Not Exactly Rocket Science” jest próbą zainteresowania nauką szerszej rzeszy czytelników poprzez unikanie żargonu i przystępną prezentację.
Mieszka w Londynie i pracuje w Cancer Research UK. Jego blog „Not Exactly Rocket Science” jest próbą zainteresowania nauką szerszej rzeszy czytelników poprzez unikanie żargonu i przystępną prezentację.